개요
핵산의 방향성
핵산(nucleic acid) 는 인산-당-염기 라는 하나의 단위가 뉴클레오타이드(nucleotide) 를 이루며, 이러한 뉴클레오타이드가 여러 개 모여서 형성된 것이다.
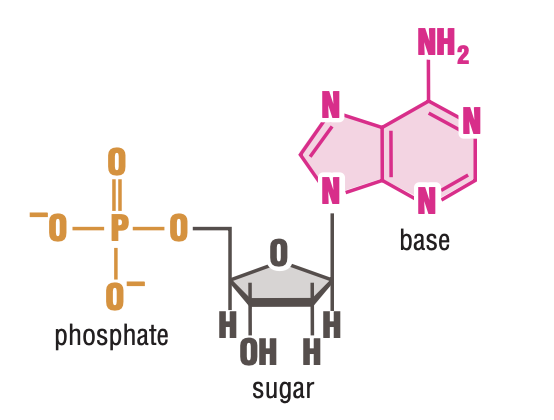
Fig 1. The Structure of the Nucleotide
즉, nucleotide는 당 분자에 의해 인산과 염기가 연결된다. 이러한 당의 종류로 Ribose 또는 Deoxyribose가 있는데, 이들은 각각 RNA 또는 DNA의 nucleotide를 구성한다. 이 두 종류의 당은 5개의 탄소 원자로 구성되어 5탄당이라 하는데, 다음과 같이 각각의 탄소에 1번부터 5번까지의 번호를 매긴다. 두 당은 그 구조와 화학식이 서로 유사하지만, Ribose에는 2’의 위치에 hydroxyl group(2’-OH)이, Deoxyribose에는 2’의 위치에 hydrogen(2’-H)이 존재한다.

Fig 2. The Types of Sugars in the RNA & DNA - Deoxyribose(left) and Ribose(right)
따라서 핵산의 방향성은 염기와 인산을 결합하는 당의 방향성에 의해 결정된다. 하나의 단일 뉴클레오타이드에서, 인산은 당의 5’에 위치한 탄소와 결합되며, 서로 다른 두 개의 뉴클레오타이드는 첫 번째 뉴클레오타이드 당의 3’ 위치에서 다음 뉴클레오타이드 당의 5’에 결합된 인산과 공유결합1한다. 그러므로, 생물학에서 핵산의 방향성을 이야기 할 때, “5’ → 3’의 방향성을 가진다” 고 말한다.

Fig 3. A single strand of DNA
핵산의 상보성
한편, 핵산에서 염기는 일반적으로2 4종류의 염기를 가진다. DNA에서는 A, T, G, C를, RNA에서는 T 대신 U를 가진다. 이러한 4종류의 염기는 DNA나 RNA가 이중 가닥을 이룰 때 서로 상보적으로 수소 결합한다. 즉, 4종류의 염기 중 2가지가 서로 짝을 이루어 수소결합을 하며, 이를 상보적 이라 한다.
수소결합은 분자를 이루는 원자 중 전기음성도가 강하고 크기가 작은 F, O, N이 인접한 수소와 분자 간 쌍극자 힘에 의해 인력을 가지는 현상을 말한다. 염기에서는 아래의 그림과 같이 질소와 산소가 수소 결합을 할 만큼 충분히 존재하며, 여러 위치 에서 수소 결합이 일어날 수 있다.
그 중 일반적인 경우에 형성되는 주요한 수소 결합을 Watson-Crick base-paring(Canonical base-paring)3이라 하며, A와 T(U)가 2개의 수소 결합을, G와 C가 3개의 수소 결합을 이룬다.
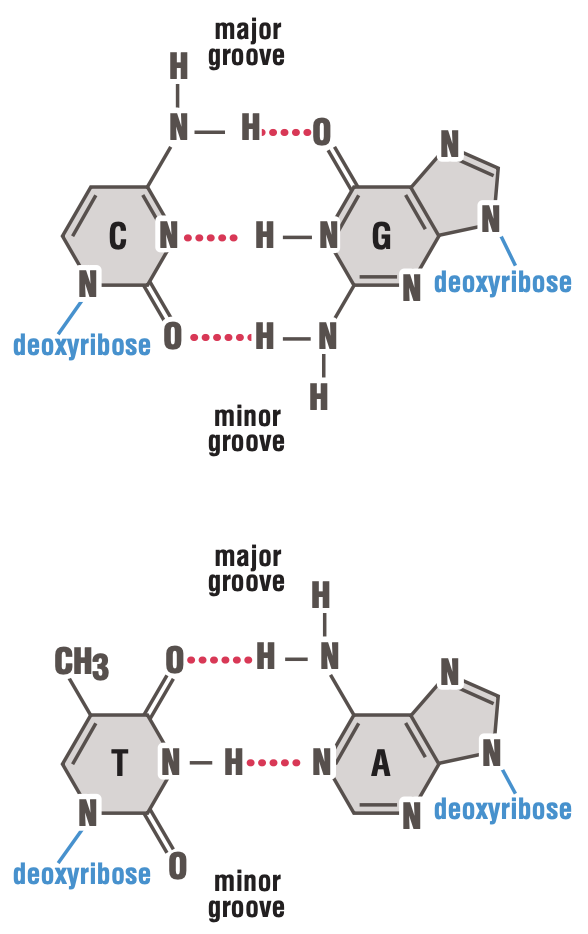
Fig 4. Watson-Crick base-paring
역상보 서열
앞선 핵산의 방향성과 상보성을 고려하여, 역상보 서열이 무엇인지 알아보도록 하자.
DNA가 이중 나선을 이룰 때, 상보성에 의해 각 가닥의 방향성이 정해진다. 즉, 어떤 한 가닥의 DNA가 이루는 방향이 5’ → 3’으로 진행된다고 할 때, 또 다른 한 가닥의 DNA 역시 5’ → 3’ 의 방향성을 가진다. 이 두 가닥이 상보성에 의해 수소결합을 이루게 되면 다음의 그림과 같이 각 가닥이 서로 반대 방향으로 진행된다.
그러므로, 우리는 어떤 임의의 서열을 읽을 때, 기준 방향을 5' → 3'으로 정하고 그 서열을 읽는다.
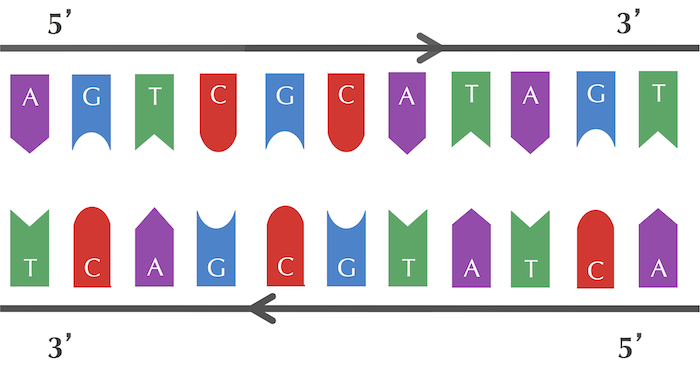
Fig 5. A complementary sequence that progresses in the reverse direction
한 가지 예시를 들어보자. 임의의 서열 “ACTGAT”가 있다고 할 때, 이 서열의 상보적인 서열은 “TGACTA” 가 된다. 그러나, 방향성을 고려하여 표시하면 다음과 같다.
5’ - ACTGAT - 3’
3’ - TGACTA - 5’
우리는 이 두 서열을 읽을 때, 주어진 5’→3’의 가닥을 주형 가닥(template strand) 이라 하고, 그 반대편의 가닥을 상보적인 가닥(complementary strand) 라 한다. 위 두 예시 서열을 기준 5’ → 3’의 방향으로 읽으면, 다음과 같이 읽을 수 있다.
5’ - ACTGAT - 3’
5’ - ATCAGT - 3’
이때, 주어진 주형 가닥에 대해 5’ → 3’ 의 방향으로 읽은 상보적인 서열을 “역상보 서열” 이라 한다.
따라서 이번 문제에서는 주어진 서열에 대한 역상보 서열을 찾아보도록 한다.
Problem
- Input: DNA 서열
- Output: 역상보 서열
- function: 주어진 DNA 서열에 대한 역상보 서열 찾기
Pseudo-code
ReverseComplement(seq)
complement = '' * |seq|
for i <- 0 to |seq|:
if seq[i] == 'A': complement[i] = 'T'
else if seq[i] == 'T': complement[i] = 'A'
else if seq[i] == 'G': complement[i] = 'C'
else if seq[i] == 'C': complement[i] = 'G'
else raise error
reverseComplement = '' * |seq|
for i <- 0 to |seq|:
reverseComplement[i] = complement[|seq| - 1 - i]
return reverseComplement
Evaluation
Time Complexity
입력 크기: $\left\vert seq \right\vert = n$
- line[2]: 상보적인 서열을 저장할 변수 complement 초기화 → $O(1)$
- line[4]: 주어진 입력 서열 seq의 모든 문자열(염기)를 순회 → $O(n)$
- line[5] ~ line[9]: 해당 순번의 염기 서열에 상응하는 상보적 서열을 complement에 저장 → $O(1)$
- line[11]: 역상보 서열을 저장할 빈 문자열 변수 reverseComplement 초기화 → $O(1)$
- line[13] ~ line[14]: 상보 서열의 모든 문자열을 순회하면서 뒤집어서 저장 → $O(n)$
- 모든 경우에 대해, 최악의 경우와 최선의 경우가 존재하지 않으며, 모든 문자열을 순회해야 correct solution이 도출됨.
- 따라서, 다음 알고리즘은 항상 일정한 시간복잡도를 가지는 알고리즘임
Total Time Complexity: $O(1) + O(n) \times O(1) + O(1) + O(n) \approxeq O(n)$
Implementation
# 주어진 서열을 상보적인 서열로 바꿔 반환하는 함수
def Complement(seq, reverse=False):
# 상보적인 서열이 어떤 것인지 저장하는 dictioinary 선언
basepair = {'A' : 'T', 'T': 'A', 'G' : 'C', 'C' : 'G'}
# 반환할 상보적 서열 문자열 변수 선언
CompSeq = ''
# 입력 서열의 각 문자 하나씩에 대한 상보적인 문자를 추가함.
# A, T, G, C 가 아닌 다른 염기에 대한 상보적인 서열은 '?'로 추가
for base in seq:
CompSeq += basepair.get(base, '?')
# reverse 매개변수를 참으로 받을 때 역상보 서열을 반환 (5' -> 3')
if reverse: return CompSeq[::-1]
# 그렇지 않으면 상보 서열만 반환 (3' -> 5')
else: return CompSeq
Discussion Points
Summary
- 임의의 DNA/RNA는 일반적인 경우 서로 상보적 인 서열을 가지고 있다.
- 각 DNA/RNA는 방향성을 가지고 있으며, 생물학에서 이를 읽는 기준 방향을 5' → 3' 으로 한다.
- 주어진 주형 가닥에 대한 상보적인 서열을 기준 방향으로 읽었을 때 역상보 서열 이라 한다.
Implementation Strategy
- 주어진 서열의 상보적인 서열을 구한다.
- 상보적인 서열을 반대 방향으로 읽는다.
Implications
- 책의 예시에서, Vibrio cholerae 의 가장 빈번한 상위 4개 9-mer가 “ATGATCAAG”, “CTTGATCAT” 임
- 이 두 서열은 서로 역상보 서열의 관계에 있으며, DnaA box를 구성할 것이라는 가설을 이끌 수 있음
- 실제로 DnaA box에 결합하는 DnaA 단백질은 DNA의 주형 가닥에 결합할 지, 상보적 가닥에 결합할 지 구분하지 않음
- 즉, 역상보 서열이 유전체에 존재하는 것은, 그 서열과 그 서열의 역상보 서열이 DnaA box를 구성할 것이라는 가설을 지지함
Reference
- Compeu, P., Pevzner, P. (2018). Bioinformatics Algorithms 3/e. 에이콘 출판사
- Craig, N., Cohen-Fix, O., Green, R., Greider, C., Storz, G., & Wolberger, C. (2010). Molecular biology: Principles of genome function. Oxford University Press.
-
이러한 공유결합을 phospho-diester bond라 한다. ↩
-
일반적이지 않은 경우, chemical modificatioin이 일어난 기타 종류의 염기를 가진다. pseudo-uridine, m6A 등의 예시가 있다. ↩
-
앞선 각주 1에서 언급된 바와 같이, chemical modification이 일어난 염기나, 기타 DNA/RNA의 구조적 특징에 의해 새로운 수소 결합의 종류가 존재한다.(Non-canonical base-pairing) ↩